
Oksidacijska fosforilacija je metabolički put koji koristi energiju oslobođenu oksidacijom hranjivih tvari za sintezu adenozin trifosfata (ATP). Gotovo svi aerobni organizmi koriste oksidacijsku fosforilaciju za proizvodnju energije, zato što je to najučinkovitiji metabolički put za proizvodnju visokoenergetskih molekula kao što je ATP. Alternativni metabolički putovi kao fermentacija ili glikoliza daleko su manje efikasni.
Tijekom oksidacijske fosforilacije, elektroni se prenose od molekula koje služe kao elektron donori na molekule elektron akceptore kao kisik u nizu redoks reakcija. Redoks reakcije oslobađaju energiju koja se koristi za sintezu ATP-a. Redoks reakcije se kod eukariota odvijaju na unutarnjoj membrani mitohondrija dok se kod prokariota odvijaju na unutarnjim staničnim membranama. Oksidacijska fosforilacija se mora uvijek odvijati na membranama jer se nastali gradijent protona između dva stanična prostora koristi kao pokretačka sila za sintezu ATP-a.
Prvi dio oksidacijske fosforilacije čini niz povezanih enzima koji nose naziv "lanac prijenosa elektrona". U eukariota taj je lanac sastavljen od pet proteinskih sustava, dok se kod prokariota može naći mnoštvo različitih enzima u raznim kombinacijama.
Energijom oslobođenom od elektrona, koji prolaze kroz navedeni enzimski sklop, dolazi do prijenosa protona kroz unutarnju mitohondrijsku membranu u procesu poznatom kao "kemiosmoza". Stvoreni gradijent protona (pH gradijent) daje potencijalnu energiju cijelom procesu koji će je postupno oslobađati protokom kroz enzim ATP sintetazu. ATP sintetaza koristi energiju stvorenu protokom protona niz međumembranski gradijent kako bi stvorila ATP reakcijom fosforilacije adenozin difosfata (ADP). To je drugi dio metaboličkog puta. Dio ATP sintetaze funkcionira kao rotacijski mehanički motor pokretan protonskim gradijentom.
Oksidacijska je fosforilacija jedan od najbitnijih dijelova metabolizma, ali ona nužno oslobađa reaktivne metabolite kisika kao superoksid (O2−) ili vodikov peroksid (H2O2) koji s lakoćom generiraju slobodne radikale. Slobodni radikali mogu teško oštetiti stanične strukture, izazvati bolesti i tijesno su povezani s procesom starenja.
Prijenos energije kod oksidacijske fosforilacije
Kod oksidacijske fosforilacije postoji sprega između dva sustava kemijskih reakcija. Reakcija koja oslobađa energiju koristi se za napajanje kemijske reakcije koje zahtijeva energiju. Jedna reakcija ne može postojati bez druge. Protok elektrona iz elektron donora kao NADH na elektron akceptore kao kisik koji se zbiva u lancu prijenosa elektrona je egzoergonska reakcija (oslobađa energiju). Sinteza ATP-a koja je endoergonska reakcija (zahtjeva energiju) dobiva je od prvog niza reakcija. Oba su sustava umetnuta u strukturu unutarnje mitohondrijske membrane. Energija oslobođena kroz lanac prijenosa elektrona koristi se za stvaranje protonskog (H+) gradijenta između dviju strana membrane u sklopu procesa kemiosmoze[1]. Povratni protok protona niz membranski gradijent daje energiju ATP sintetazi za proizvodnju ATP-a.[2]
Količina oslobođene energije oksidacijskom fosforilacijom vrlo je velika u odnosu na druge metaboličke putove koji stvaraju energiju. Degradacijom jedne molekule glukoze kod glikolize proizvedu se samo 2 molekule ATP-a. Ista molekula glukoze daje 10 molekula NADH i 2 molekule sukcinata tokom Krebsovog ciklusa, a koje za vrijeme oksidacijske fosforilacije daju 30 do 36 molekula ATP-a[3]. Svaki ciklus beta oksidacije masnih kiselina daje oko 14 molekula ATP-a. U stvarnosti, proizvodnja ATP-a uvijek je nešto niža od teoretskih maksimalnih vrijednosti jer se se manja količina protona gubi spontanim prolazom kroz mitohondrijsku membranu niz gradijent[4].
Molekule prijenosnici protona i elektrona

Lanac prijenosa elektrona prenosi elektrone prebacujući ih od elektron donora na elektron akceptore dok se protoni prebacuju kroz mitohondrijsku membranu. U oba slučaja prijenos vrše molekule koje mogu biti slobodne u otopini ili vezane za enzime. U međumembranskom prostoru mitohondrija elektrone prenosi bjelančevina citokrom c koja je hidrofilna (topljiva u vodi)[5]. Citokrom c prenosi samo elektrone prenesenih putem redukcija i oksidacija atoma željeza unutar hem grupe koja se nalazi u građi same bjelančevine kao prostetska skupina. Kod bakterija se citokrom c nalazi u periplazmatskom prostoru.[6]
U kontekstu unutarnje mitohondrijske membrane nalazi se koenzim Q10 (Q) ili ubikinon. Ova je molekula iz skupine benzokinona vrlo lipofilna (topiva u mastima) i može prenositi protone i elektrone putem redoks reakcija [7]. Njena joj lipofilna priroda omogućuje da se slobodno kreće unutar same membrane. Prihvaćenjem dva elektrona i dva protona za vrijeme Krebsovog ciklusa ubikinon (Q) prelazi u reducirani oblik ubikinol (QH2) dok se oksidacijom za vrijeme oksidacijske fosforilacije vraća u prvobitno stanje (Q). Budući da se enzimi koji reduciraju ubikinon nalaze na jednoj strani membrane, a enzimi koji oksidiraju ubikinol na drugoj, ova molekula zapravo ima ulogu povezivanja dviju reakcija i transporta protona kroz samu membranu[8]. Kod bakterija prijenos se vrši putem metakinona i ubikinona[9].
U sklopu samih enzima nalazimo različite molekule koje prenose elektrone: flavini, molekularni sustavi željezo-sumpor i citokromi[2][10]. Sustavi željezo-sumpor su najčešći i ima ih više vrsta. Najjednostavnija vrsta u lancu prijenosa elektrona je sklop dva atoma željeza s dva atoma sumpora koji nosi naziv [2Fe–2S] sklop (cluster). Slični sklop naziva [4Fe–4S] prostorno je složen u obliku kocke i sadrži četiri atoma željeza u paru s četiri atoma sumpora koji su dio molekule cisteina. Navedeni kofaktori koji sadrže ione metala ulaze i izlaze iz redoks reakcija bez vezanja i oslobađanja protona, oni jednostavno generiraju stalni protok elektrona iz jednog enzimskog sustava na drugi[11]. Elektroni se tako kreću putem kvantnog fenomena "#tunel efekta" koji je vrlo brz na udaljenostima manjim od 1.4×10−9 m[12].
Lanac prijenosa elektrona kod eukariota
Nekoliko bitnih metaboličkih putova, kao glikoliza, Krebsov ciklus i beta oksidacija proizvode reducirani kofaktor NADH koji sadrži elektrone visokog potencijala oslobađanja. NADH je stoga u stanju osloboditi veliku količinu energije za vrijeme oksidacije. Stanica ne oslobađa odjednom svu energiju prisutnu u NADH, već se elektroni postupno oslobađaju iz reduciranog kofaktora i polako prelaze na kisik kao oksidativno sredstvo putem enizma koji čine lanac prijenosa elektrona. Enzimi prenositelji elektrona tako oslobađaju energiju vrlo postupno, povećavajući učinkovitost cijelog procesa. Taj sustav enzima nosi naziv "lanac prijenosa elektrona" i nalazi se u sklopu unutarnje mitohondrijske membrane. Pojedini enzimi navedenoga enzimskog lanca numerirani su rimskim brojevima od kompleksa I do kompleksa IV.
Kod eukariota, enzimi koriste energiju oslobođenu kod oksidacije NADH kroz sustav prijenosa elektrona za prijenos protona (H+) kroz unutarnju mitohondrijsku membranu. Nakupljanje veće količine protona u međumembranskom prostoru stvara elektrokemijski gradijent kroz samu membranu. Energiju pohranjenu u gradijentu protona koristi enzim ATP sintetaza kako bi kontinuirano proizvodila ATP.
| Enzim | Redoks par | Standardni elektrodni potencijal
(Volti) |
|---|---|---|
| NADH dehidrogenaza | NAD+ / NADH | −0,32[13] |
| sukcinat dehidrogenaza | flavin mononukleotid (FMN) ili FAD / FMNH2 ili FADH2 | −0,20[13] |
| koenzim Q - citokrom c reduktaza ili citokrom bc1 sustav | koenzim Q10oksidiran / koenzim Q10reduciran | +0,06[13] |
| citokrom bc1 sustav | citokrom boksidiran / citokrom breduciran | +0,12[13] |
| citokrom c oksidaza ili kompleks IV | citokrom coksidiran / citokrom creduciran | +0,22[13] |
| kompleks IV | citokrom aoksidiran / citokrom areduciran | +0,29[13] |
| kompleks IV | O2 / HO− | +0,82[13] |
| Uvjeti: pH = 7[13] | ||
NADH-koenzim Q oksidoreduktaza ((kompleks I)

NADH-koenzim Q oksidoreduktaza ili NADH dehidrogenaza (kompleks I) je prvi enzim lanca prijenosa elektrona[14]. Radi se o vrlo velikom enzimu, molekularne mase od 1000 kilodaltona (KDa) koji se sastoji od 46 podjedinica[15]. Detaljna struktura je prvenstveno poznata iz kristalografskih studija bakterijske NADH dehidrogenaze[16][17]; oblikom podsjeća na obrnuto slovo "L", gdje se jedan od krakova nalazi u kontekstu unutarnje mitohondrijske membrane, dok drugi krak strši prema matriksu mitohondrija[18][19]. Geni koji dekodiraju pojedine podjedinice kompleksa I nalaze se i u staničnoj jezgri i u mitohondrijskom genomu, što je prilično uobičajeno za mitohondrijske enzime.
Reakcija koju katalizira kompleks I sastoji se od redukcije koenzima Q10 (ubikinona) od strane NADH, prijenosom dva elektrona. Koenzim Q10 ili ubikinon je označen u jednadžbi niže kao Q:
Početak kemijske reakcije i cijelog lanca prijenosa elektrona je vezanje jedne molekule NADH za kompleks I i donacija dva elektrona i jednog protona kofaktoru flavin mononukleotidu (FMN), koji predstavlja prvu prostetičku grupu samog enzimskog kompleksa. FMN prelazi u reducirani oblik FMNH2 primanjem dva elektrona i jednog protona od NADH, dok drugi proton prima direktno iz matriksa. Elektroni su potom prebačeni nizom Fe-S sklopova (clusters), koji predstavljaju drugu prostetičku grupu enzimskog kompleksa. Kompleks I sadrži obje vrste sklopova željezo-sumpor: [2Fe–2S] i [4Fe–4S][20].
Proces prijenosa elektrona je nepovratan, jer elektroni postupno gube potencijal za 360 mV što je ekvivalentno ΔG=-16.6 Kcal/mol. Oslobođena energija omogućava prebacivanje četiri protona (H+) iz matriksa u međumembranski prostor, promjenom konformacije samog kompleksa I. Elektroni se na kraju reakcije prebacuju iz Fe-S sklopova na molekulu ubikinona koja se nalazi unutar mitohondrijske membrane. Osim primanja elektrona, redukcija ubikinona (Q) predviđa prijenos dva protona iz matriksa na molekulu generirajući ubikinol (QH2), povećavajući tako gradijent protona između matriksa i međumembranskog prostora.
Sukcinat-Koenzim Q oksidoreduktaza (kompleks II)

Sukcinat-Koenzim Q oksidoreduktaza (Kompleks II) ili sukcinat dehidrogenaza predstavlja drugi ulaz u lanac prijenosa elektrona[21]. Ovo je jedini enzim koji ulazi u sastav i Krebsovog ciklusa i oksidacijske fosforilacije. Struktura kompleksa II predviđa četiri proteinske podjedinice na koje su vezane različite prostetičke grupe: flavin adenin dinukleotid (FAD), Fe-S sklopovi i molekula hema koja nema ulogu prijenosa elektrona na koenzim Q, već se pretpostavlja da smanjuje koncentraciju potencijalno štetnih reaktivnih vrsta kisika[22][23].
Enzim katalizira oksidaciju sukcinata u fumarat i redukciju ubikinona u ubikinol. Ova reakcija oslobađa manje energije od oksidacije NADH, tako da kompleks II ne prebacuje direktno protone u međumembranski prostor.
Flavoprotein-Koenzim Q oksidoreduktaza
Flavoprotein-Koenzim Q oksidoreduktaza (ETF-Q oksidoreduktaza) ili Flavoprotein dehidrogenaza predstavlja treću ulaznu točku u lanac prijenosa elektrona. Ovaj enzim prima elektrone od flavoproteina i prebacuje ih na ubikinon (Q10) kako bi izvršio redukciju[24]. ETF-Q oksidoreduktaza sadrži flavin kao prostetičku grupu i jedan [4Fe–4S] sklop[25]. Za razliku od drugih enzima, ETF-Q oksidoreduktaza se ne nalazi u kontekstu mitohondrijske membrane, već je smješten na samoj površini membrane na strani mitohondrijskog matriksa.
Ovaj je enzim kod sisavaca važan u metaboličkom putu beta oksidacije masnih kiselina, katabolizmu nekih amino kiselina i degradaciji kolina, jer ima ulogu primanja elektrona od više acetil CoA dehidrogenaza[26][27]. Njegova je funkcija važna i kod biljaka, jer ulazi u metaboličke procese koje omogućavaju biljkama preživljavanje kod produženih perioda manjka svjetla[28].
Koenzim Q-Citokrom c oksidoreduktaza (kompleks III)

Enzimski kompleks koenzim Q-citokrom c oksidoreduktaza (citokrom c reduktaza ili citokrom bc1 kompleks ili kompleks III) se kod sisavaca pojavljuje kao dimer[29][30]. Svaka je polovica sastavljena od 11 proteinskih podjedinica, jednog [2Fe-2S] željezo-sumpor sklopa i tri citokroma od kojih jedan citokrom c1 i dva citokroma b[31]. Citokrom je vrsta bjelančevine labavo pridružena mitohondriju unutar međumembranskog prostora, koja sadrži hem kao prostetičku grupu i ima ulogu prijenosa elektrona iz stanja visoke energije, postupno u stanje niže energije. Atomi željeza unutar hema sposobni su prelaziti iz reduciranog stanja (Fe+2) u oksidirano stanje (Fe+3) u tijeku prolaza elektrona kroz bjelančevinu.
Reakcija koju katalizira kompleks III je oksidacija jedne molekule ubikinola i redukcija dviju molekula citokroma c. Za razliku od koenzima Q, koji prenosi dva elektrona, citokrom c prenosi samo jedan elektron.
Budući da je u jednom trenutku moguć prijenos samo jednog od elektrona s QH2 donora na citokrom akceptor, mehanizam prijenosa elektrona kod kompleksa III je nešto složeniji nego kod ostalih kompleksa lanca prijenosa elektrona. Proces se zbiva u dva koraka i nosi naziv ciklus Q[32]. U prvom koraku enzim veže na sebe tri supstrata. Prvi je reducirani kofaktor QH2 koji oksidacijom prebacuje jedan elektron na drugi supstrat citokrom c i oslobađa dva protona (H+) u međumembranski prostor. Treći supstrat je oksidirani koenzim Q, koji prihvaća drugi elektron iz QH2 i prelazi u reducirani oblik Q-, koji nosi naziv slobodni radikal ubisemikinon. Prva se dva supstrata oslobađaju, dok međuspoj ubisemikinon ostaje vezan za kompleks III. U drugom koraku ciklusa Q, druga molekula QH2 veže se na kompleks III i ponovo prebacuje prvi elektron na citokrom c. Drugi se elektron sada prebacuje na vezani ubisemikinon (Q-), koji se reducira u QH2 primanjem dva protona iz mitohondrijskog matriksa. Reducirani QH2 se tada oslobađa od veze s kompleksom III[33].
Za napomenuti je da se koenzim Q reducira u ubikinol na unutarnjoj strani mitohondrijske membrane (strana matriksa), a oksidira na vanjskoj strani (strana međumembranskog prostora), tako da dolazi do prijenosa protona kroz membranu pojačavajući tako protonski gradijent[2]. Ciklus Q je vrlo važan proces, jer povećava učinkovitost prijenosa protona kroz membranu. Kad bi proces koristio jednu molekulu QH2 da izravno reducira dva citokroma c, učinkovitost bi se prepolovila sa samo jednim prenesenim protonom za svaki reducirani citokrom c [2].
Citokrom c oksidaza (kompleks IV)

Citokrom c oksidaza ili kompleks IV je posljednji multienzimski kompleks u u nizu koji čini lanac prijenosa elektrona[34]. Ovaj enzim ima, kod sisavaca, izuzetno složenu građu od 13 podjednica, dva hema kao prostetičku grupu, i više kofaktora koji sadrže ione metala; tri atoma bakra, jedan atom magnezija i jedan cinka[35].
Ova oksidaza katalizira posljednju reakciju u lancu prijenosa elektrona prenoseći elektrone na kisik, dok istovremeno pumpa protone kroz membranu[36]. Kisik ima ulogu krajnjeg primatelja elektrona i biva reduciran u vodu, koja je jedan od terminalnih produkata oksidacijske fosforilacije. Vodik potreban za redukciju kisika u vodu dolazi iz matriksa mitohondrija smanjujući tako njegovu koncentraciju u samom matriksu, što povećava protonski gradijent potreban za dalju sintezu ATP. Jednadžba reakcije oksidacije citokroma c:
Ostale reduktaze i oksidaze
U mnogih se eukariota lanac prijenosa elektona bitno razlikuje od onog gore opisanog karakterističnog za sisavce i ostale složenije organizme. Biljke npr. imaju NADH oksidaze, koje vrše oksidaciju NADH u citosolu umjesto u mitohondrijskom matriksu i prenose elektrone izravno na ubikinon[37]. Ovi enzimi ne prenose protone, stoga reduciraju ubikinon bez promjene u elektrokemijskom gradijentu između dvije strane membrane[38].
Drugi primjer različitog lanca prijenosa elektrona su alternativne oksidaze prisutne kod biljaka, gljiva, protista i vjerojatno nekih životinja.[39][40]. Ovi enzimi izravno prenose elektrone iz ubikinola na kisik[41].
Opisani alternativni metabolički putovi daju manji prinos pri proizvodnji ATP u odnosu na puni put oksidacijske fosforilacije. Korist skraćenih metaboličkih putova nije u potpunosti jasna. Alternativne se oksidaze proizvode kao odgovor na biološki stres kao hladnoću, reaktivne vrste kisika, zaraze i ostale čimbenike koji inhibiraju lanac prijenosa elektrona[42][43]. Ovi alternativni metabolički putovi vjerojatno predstavljaju prilagodbu, koja povećava otpornost organizma na neke vanjske štetne faktore[44].
Prostorna organizacija enzimskih kompleksa
U prošlosti se pretpostavljalo da se enzimi koji čine lanac prijenosa elektrona mogu slobodno kretati unutar mitohondrijske membrane nezavisno jedan od drugoga[45]. Novija istraživanja su pak dokazala da se enzimski kompleksi povezuju u strukture višeg reda međusobno interagirajućih enzima koji nose naziv superkompleksi ili "respirasomi"[46][47]. Ovo udruživanje omogućuje kanaliziranje supstrata između različitih enzimskih sustava, povećavajući tako brzinu i učinkovitost prijenosa elektrona[48]. Omjer različitih enzima unutar superkompleksa kod sisavaca može biti različit. Neke studije daju naznake da bi omjer kompleksa I/II/III/IV i ATP sintetaze mogao biti 1:1:3:7:4[49], ali ovi podaci još nisu potpuno potvrđeni[15][50].
Lanac prijenosa elektrona kod prokariota
Za razliku od eukariota, kod kojih su enzimi u sustavu prijenosa vrlo slični, prokarioti (bakterije i arheobakterije) posjeduju širok spektar različitih enzima prijenosnika elektrona i isto tako i supstrata i kofaktora[51]. Ono što je zajedničko s eukariotima je to da se oslobođena energija tijekom prijenosa elektrona za oksidacije supstrata koristi za pumpanje iona kroz staničnu membranu i generiranje elektrokemijskog gradijenta. Najbolje izučen primjer oksidacijske fosforilacije je onaj kod Escherichia coli, dok je proces kod arheobakterija[52] malo poznat.
Osnovna razlika između oksidacijske fosforilacije u eukariota i prokariota je ta da prokarioti mogu koristiti mnoštvo različitih spojeva kao elektron donore i akceptore, što im omogućava preživljavanje u različitim okolišnim uvjetima[53]. Escherichia coli može koristiti velik broj različitih redukcijskih i oksidacijskih parova, koji su navedeni u tablici niže. Standardni elektrodni potencija svakog kemijskog spoja daje mjeru oslobođene energije za vrijeme redukcije ili oksidacije. Reduktivni spojevi imaju negativni potencijal, dok kod oksidativnih spojeva potencijal je pozitivan. Što je veća razlika u standardnom elektrodnom potencijalu između reduktivnog i oksidativnog spoja, to će se više energije osloboditi prilikom reakcije.
| Enzim | Redoks par | Standardni elektrodni potencijal
(Volti) |
|---|---|---|
| format dehidrogenaza | bikarbonat / format | |
| hidrogenaza | proton / vodik | |
| NADH dehidrogenaza | NAD+ / NADH | |
| glicerol-3-fosfat dehidrogenaza | dihdroksiaceton fosfat (DHAP) / glicerol 3-fosfat (glicerol-3-P) | |
| piruvat dehidrogenaza-piruvat oksidaza | octena kiselina (acetat) + ugljikov (IV) oksid (CO2)/ piruvat | |
| laktat dehdrogenaza | piruvat / mliječna kiselina (laktat) | |
| D-amino kiselina dehidrogenza | 2-oksokiselina + amonijak / D-amino kiselina | |
| kinoprotein glukoza dehidrogenaza | glukonat / glukoza | |
| sukcinat-koenzim Q reduktaza (sukcinat dehidrogenaza) | fumarat / sukcinat | |
| ubikinol oksidaza | kisik / voda | |
| nitrat reduktaza | nitrat / nitrit | |
| nitrit reduktaza | nitrit / amonijak | |
| dimetil sulfoksid reduktaza | dimetil sulfoksid (DMSO) / dimetil sulfid (DMS) | |
| trimetilamin N-oksid reduktaza | trimetilamin N-oksid (TMAO) / trimetilamin (TMA) | |
| fumarat reduktaza | fumarat / sukcinat |
Prokarioti mogu kontrolirati korištenje različitih elektron donora i akceptora mijenjajući ekspresiju različitih enzima koji ulaze u lanac prijenosa elektrona, kao odgovor na promjene vanjskih uvjeta[55]. Ova prilagodljivost je moguća jer različiti enzimi koriste ubikinon kao univerzalni kofaktor. Različite varijante lanca prijenosa elektrona su modularno organizirane, tako da se enzimski sustavi mogu s lakoćom mijenjati kao odgovor na promjene u okolišu.
Osim opisane metaboličke raznolikosti, prokarioti posjeduju mnoštvo izoenzima, različitih enzima koji kataliziraju istu reakciju. Kod Escherichia coli postoje dvije varijante ubikinol oksidaze koje koriste kisik kao elektron akceptor. U uvjetima velike koncentracije kisika, bakterija koristi oksidazu s niskim afinitetom prema kisiku koja može prenijeti dva protona po svakom elektronu, dok u uvjetima niske koncentracije kisika, sustav se prebacuje na oksidazu koja prenosi samo jedan proton po elektronu, ali ima visok afinitet prema kisiku[56].
ATP sintetaza (kompleks V)
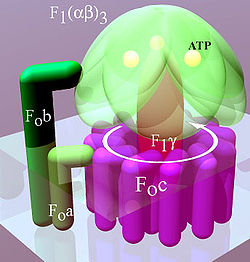
ATP sintetaza ili kompleks V, predstavlja posljednji enzim u metaboličkom putu oksidacijske fosforilacije. Pronađen je u svim živim organizmima i smatra se da je jedan od najvažnijih enzima za održavanje života uopće. Njegov je način funkcioniranja jednak i kod prokariota i kod eukariota[57]. ATP sintetaza koristi elektrokemijsku energiju pohranjenu u protonskom gradijentu između dviju strana membrane kako bi katalizirao sintezu ATP iz ADP i fosfata. Procijenjeno je da su potrebna tri do četiri protona za sintezu jedne molekule ATP[58][59], ali stanice mogu mijenjati ovaj omjer kako bi se prilagodile različitim okolišnim uvjetima[60].
Reakcija fosforilacije je povratna kemijska reakcija u ravnoteži, može se stoga okrenuti ako se okrene protonski gradijent. Bez energije iz protonskog gradijenta, reakcija bi tekla od desna u lijevo; enzim bi razgrađivao ATP i pumpao protone kroz membranu iz matriksa u međumembranski prostor. U prisutnosti jakog protonskog gradijenta, kemijska reakcija teče u suprotnom smjeru, od lijeva na desno, dopušta protonima da teku niz gradijent i proizvodi ATP iz ADP[57]. U nekim slučajevima isti ili slični enzim H+ ATPaza ili tzv. protonska pumpa, troši energiju iz hidrolize ATP kako bi prenijela protone u vakuole i povećala tako kiselost (smanjila pH)[61]
. Ova je reakcija odgovorna za proizvodnju kiselosti u želudčanom sekretu.
ATP sintetaza je veliki proteinski sklop, koji oblikom ugrubo podsjeća na gljivu (vidi sliku). Ovaj je kompleks kod sisavaca sastavljen od dva osnovna dijela: jednog integriranog u membranu FO i drugog perifernog dijela F1. Cijeli sustav ima 16 podjedinica, ukupne molekularne mase 600 kilodaltona (KDa)[62]. Dio enzima pozicioniran u debljini same membrane nosi naziv FO i sastavljen je od jedne podjedinice a, dviju podjedinica b i 10 kružno raspoređenih podjedinica c, koje formiraju kanal kroz kojeg prolaze protoni. Drugi dio enzimskog sustava naziva se F1 i čini ga sferični vanjski dio i središnja podjedinica γ u obliku štapića koja je na vrhu spojena s vanjskim dijelom i cijelom svojom dužinom zauzima protonski kanal načinjen od podjedinica c. Kuglasta struktura na kraju F1 dijela sastavljena je od šest bjelančevina dvije različite vrste: tri α podjedinice i tri β podjedinice organizirane u tri dimera α-β, slično narančinim kriškama. U tom dijelu ATP sintetaze dolazi do sinteze ATP[63]. Obje su vrste podjedinica sposobne vezati nukleotide, ali samo β jedinica katalizira reakciju sinteze ATP.
Tijekom protoka protona kroz FO membranski kanal, podjedinice c rotiraju oko središnje osi[64]. Rotacija vjerojatno nastaje kao posljedica ionizacije aminokiselina u prstenu sastavljenoga od c podjedinica (c-prsten), koje uzrokuju elektrokemijske interakcije i pokreću tako sam prsten[65]. Budući da je c-prsten čvrsto vezan za središnju F1 γ podjedinicu, rotacija uzrokuje vrtnju vrha γ podjedinice između α-β dimera periferne F1 kuglaste strukture. F1 kuglasta struktura se pritom ne okreće, jer je blokirana podjedinicama F0b. Vrtnja vrha centralne podjedinice γ osigurava potrebnu energiju aktivnim mjestima na β podjedinicama koje naizmjence kataliziraju sintezu i oslobođanje ATP[66].
Rotacijska kataliza ATP

Dio ATP sintetaze odgovoran za sintezu ATP je kuglasta F1 struktura sastavljena od α-β dimera. Na β podjedinicama nalaze se aktivna mjesta koja naizmjence vrše sintezu ATP: jedan se nalazi u konformaciji β-ATP i drži vezan ATP, drugi se nalazi u konformaciji β-ADP i sposoban je vezati supstrat ADP i treći ima aktivno mjesto β-prazno i nije sposoban vezati supstrate.[67]. Rotacijska sila uzrokovana protokom protona niz gradijent pokreće rotaciju centralne osi (podjedinica γ) koja ulazi u dodir s beta podjedinicama.
Posljedica je konformacijska promjena kod koje se aktivno mjesto β-ATP mijenja u β-prazno ispuštajući ATP iz prethodne rotacije (tamno crveno u slici). Potom mjesto β-prazno prelazi u konformaciju β-ADP i labavo veže ADP i fosfat iz otopine u matriksu (crveno), da bi na kraju aktivno mjesto prešlo u konformaciju β-ATP gdje dolazi do kondenzacije ADP i fosfata u ATP (ružičasto). Ovaj se ciklus neprestano ponavlja i dobiva naziv rotacijska kataliza.
Kod nekih bakterija i arheobakterija kao Metanococcus, sinteza ATP je uzrokovana protokom iona natrija a ne protona[68][69]. Neki od ovih organizama posjeduju A1AO sintetazu[70] (umjesto F1FO), oblik enzima s vrlo malo sličnosti s podjedinicama eukariotske ATP sintetaze, koja bi mogla biti u stanju koristiti Na+ ione umjesto H+.
Reaktivne vrste kisika
Molekularni kisik (O2) je idelani akceptor elektrona, zahvaljujući svom vrlo jakom oksidacijskom potencijalu, ali redukcija kisika može proizvesti potencijalno štetne međuspojeve[71]. Prijenos četiri elektrona i četiri protona na molekulu kisika reducira ga u bezopasnu molekulu vode, dok prijenosom jednog ili dva elektrona stvara se visoko reaktivni anion superoksid ili peroksid:
Navedeni reaktivni međuspojevi kisika i produkti njihovih reakcija, kao hidroksilni radikal, mogu biti vrlo štetni za stanične strukture jer mogu oksidirati stanične bjelančevine i lipide i uzrokovati mutacije u DNK. Pretjerana oštećenja nastala nekontroliranom oksidacijom unutarnjih struktura mogu dovesti do apoptoze, programirane smrti koju stanica sama započne u slučaju da su oštećenja nemoguća za popravak, a vjerojatno su i jedan od uzroka biološkog starenja[72][73].
Enzimski sustav citokrom c oksidaza (kompleks IV) izuzetno je učinkovit u redukciji kisika u vodu i ispušta vrlo male količine djelomično reduciranih međuspojeva, međutim lanac prijenosa elektrona neprekidno ispušta superoksid i peroksid ione[74]. Posebno je osjetljiv korak redukcija koenzima Q od strane kompleksa III, jer podrazumijeva stvaranje visoko reaktivnog slobodnog radikala ubisemikinona kao međuspoja u Q ciklusu što može pridonijeti stvaranju superoksid aniona[75]. Veća aktivnost lanca prijenosa elektrona stvara veći protonski gradijent između dviju strana mitohondrijske membrane, time i veću proizvodnju ATP, ali kao nuspojavu i veću proizvodnju reaktivnih vrsta kisika. Postoje naznake da mitohondriji reguliraju intenzitet aktivnosti mehanizmom negativne povratne sprege kako bi uravnotežili proizvodnju ATP s negativnom nuspojavom proizvodnje oksidanata[76][77].
Kako bi neutralizirali reaktivne vrste kisika, stanice koriste različite antioksidacijske sustave, kao vitamin C i E koji imaju direktno antioksidacijsko djelovanje i enzime superoksid dizmutaza, katalaza i peroksidaza koje degradiraju reaktivne međuspojeve svodeći tako na minimum njihovo štetno djelovanje[71].
Inhibitori
Poznato je nekoliko vrsta kemijskih spojeva koji inhibiraju metabolički put oksidacijske fosforilacije na točno određenim točkama. U tablici niže navedeni su najpoznatiji lijekovi i otrovi koji djeluju na oksidacijsku fosforilaciju:
| Kemijski spoj | Priroda spoja | Utjecaj na oksidacijsku fosforilaciju |
|---|---|---|
| cijanid ugljični monoksid (CO) azid |
otrovi | Inhibiraju lanac prijenosa elektrona, vezivanjem na Fe–Cu centar citokrom c oksidaze puno jače od kisika, sprječavajući tako njegovu redukciju.[78] |
| oligomicin | antibiotik | Inhibira ATP sitetazu tako da blokira protok protona kroz Fo podjedinicu.[79] |
| karbonil cijanid m-klorofenil hidrazon (CCCP) 2,4-dinitrofenol |
otrovi | Ionofori koji neutraliziraju protonski gradijent tako što prenose protone kroz membranu. [80] |
| rotenon | pesticid | Sprječava prijenos elektrona od kompleksa I na ubikinon tako da blokira aktivno mjesto vezanja ubikinona.[81] |
| malonat i oksaloacetat | metabolit | Kompetitivni inhibitori sukcinat dehidrogenaze (kompleks II).[82] |
Toksična se aktivnost svakog od navedenih inhibitora ne ograničava samo na oksidacijsku fosforilaciju, već se reflektira na cijeli stanični metabolizam. Blokada oksidacijske fosforilacije onemogućava ponovnu oksidaciju kofaktora NADH i FADH2 u NAD+ i FAD, što oduzima osnovne supstrate Krebsovom ciklusu i glikolizi što ima kao posljedicu njihovo postupno gašenje.
Onesposobljavanje pojedinih dijelova oksidacijske fosforilacije ne mora uvijek biti štetno. U smeđem masnom tkivu postoje protonski kanali koji kontrolirano disociraju lanac prijenosa elektrona od sinteze ATP, što proizvodi toplinu. Ovaj je proces posebno važan za održavanje tjelesne temperature kod životinja koje zimi idu u hibernaciju ("zimski san")[83].
Povijest otkrića
Rasvjetljavanje metaboličkog puta oksidacijske fosforilacije počinje još 1906. kad je Artur Harden opisao važnost fosfata u staničnoj fermentaciji[84]. 1940. danski biokemičar Herman Kalkar otkriva vezu između oksidacije šećera i proizvodnje ATP[85], a 1949. Morris Friedkin i Albert L. Lehninger dokazuju da koenzim NADH povezuje različite metaboličke putove kao Krebsov ciklus i sintezu ATP[86].
Mehanizam proizvodnje ATP u stanici ostaje nepoznat dvadesetak godina. U tom su se periodu znanstvenici usredotočili na potrage hipotetskog "visokoenergetskog međuspoja" koji povezuje oksidaciju s fosforilacijom[87]. Mehanizam je objašnjen 1961. kad je britanski biokemičar Peter D. Mitchell objavio svoju "kemiosmotičku teoriju"[88]. U početku je kemiosmoza naišla na žestok otpor u akademskim krugovima, ali je poslije postupno prihvaćena i dodatno potvrđena a Michellu je dodijeljena Nobelova nagrada za svoje otkriće 1978.[89][90]
Kasnija su se istraživanja usredotočila na izolaciju i proučavanje pojedinih enzima. Posebno važan korak u razumijevanju oksidacijske fosforilacije bilo je objašnjenje mehanizma funkcioniranja ATP sintetaze rotacijskom katalizom[67][91] za što su John E. Walker i Paul D. Boyer dobili Nobelovu nagradu 1997.[92]
Izvori
- ↑ Mitchell P, Moyle J (1967). "Chemiosmotic hypothesis of oxidative phosphorylation". Nature 213 (5072): 137–9. doi:10.1038/213137a0. PMID 4291593
- ↑ 2,0 2,1 2,2 2,3 Schultz B, Chan S (2001). "Structures and proton-pumping strategies of mitochondrial respiratory enzymes". Annu Rev Biophys Biomol Struct 30: 23–65. doi:10.1146/annurev.biophys.30.1.23. PMID 11340051
- ↑ Rich PR (2003). "The molecular machinery of Keilin's respiratory chain". Biochem. Soc. Trans. 31 (Pt 6): 1095–105. doi:10.1042/BST0311095. PMID 14641005. http://www.biochemsoctrans.org/bst/031/1095/bst0311095.htm
- ↑ Porter RK, Brand MD (1995). "Mitochondrial proton conductance and H+/O ratio are independent of electron transport rate in isolated hepatocytes". Biochem. J. 310 ((Pt 2)): 379–82. PMC 1135905. PMID 7654171. //www.ncbi.nlm.nih.gov/pmc/articles/PMC1135905/
- ↑ Mathews FS (1985). "The structure, function and evolution of cytochromes". Prog. Biophys. Mol. Biol. 45 (1): 1–56. doi:10.1016/0079-6107(85)90004-5. PMID 3881803
- ↑ Wood PM (1983). "Why do c-type cytochromes exist?". FEBS Lett. 164 (2): 223–6. doi:10.1016/0014-5793(83)80289-0. PMID 6317447
- ↑ Crane FL (1 prosinac 2001). "Biochemical functions of coenzyme Q10". Journal of the American College of Nutrition 20 (6): 591–8. PMID 11771674. http://www.jacn.org/cgi/content/full/20/6/591
- ↑ Mitchell P (1979). "Keilin's respiratory chain concept and its chemiosmotic consequences". Science 206 (4423): 1148–59. doi:10.1126/science.388618. PMID 388618
- ↑ Søballe B, Poole RK (1999). "Microbial ubiquinones: multiple roles in respiration, gene regulation and oxidative stress management" (PDF). Microbiology (Reading, Engl.) 145 (Pt 8): 1817–30. PMID 10463148. http://mic.sgmjournals.org/cgi/reprint/145/8/1817.pdf
- ↑ Johnson D, Dean D, Smith A, Johnson M (2005). "Structure, function, and formation of biological iron-sulfur clusters". Annu Rev Biochem 74: 247–81. doi:10.1146/annurev.biochem.74.082803.133518. PMID 15952888
- ↑ Page CC, Moser CC, Chen X, Dutton PL (1999). "Natural engineering principles of electron tunnelling in biological oxidation-reduction". Nature 402 (6757): 47–52. doi:10.1038/46972. PMID 10573417
- ↑ Leys D, Scrutton NS (2004). "Electrical circuitry in biology: emerging principles from protein structure". Curr. Opin. Struct. Biol. 14 (6): 642–7. doi:10.1016/j.sbi.2004.10.002. PMID 15582386
- ↑ 13,0 13,1 13,2 13,3 13,4 13,5 13,6 13,7 Medical CHEMISTRY Compendium. By Anders Overgaard Pedersen and Henning Nielsen. Aarhus University. 2008
- ↑ Hirst J (2005.). "Energy transduction by respiratory complex I—an evaluation of current knowledge" (PDF). Biochem. Soc. Trans. 33 (Pt 3): 525–9. doi:10.1042/BST0330525. PMID 15916556. http://www.biochemsoctrans.org/bst/033/0525/0330525.pdf
- ↑ 15,0 15,1 Lenaz G, Fato R, Genova M, Bergamini C, Bianchi C, Biondi A (2006). "Mitochondrial Complex I: structural and functional aspects". Biochim Biophys Acta 1757 (9–10): 1406–20. doi:10.1016/j.bbabio.2006.05.007. PMID 16828051
- ↑ Sazanov L.A., Hinchliffe P. (2006.). "Structure of the hydrophilic domain of respiratory complex I from Thermus thermophilus". Science 311 (5766): 1430–1436. doi:10.1126/science.1123809. PMID 16469879
- ↑ Efremov R.G., Baradaran R., & Sazanov L.A., (2010) The arcdhitecture of respiratory complex I, Nature 465, 441-445
- ↑ Baranova EA, Holt PJ, Sazanov LA (2007.). "Projection structure of the membrane domain of Escherichia coli respiratory complex I at 8 A resolution". J. Mol. Biol. 366 (1): 140–54. doi:10.1016/j.jmb.2006.11.026. PMID 17157874
- ↑ Friedrich T, Böttcher B (2004). "The gross structure of the respiratory complex I: a Lego System". Biochim. Biophys. Acta 1608 (1): 1–9. doi:10.1016/j.bbabio.2003.10.002. PMID 14741580
- ↑ Hirst J (siječanj 2010.). "Towards the molecular mechanism of respiratory complex I". Biochem. J. 425 (2): 327–39. doi:10.1042/BJ20091382. PMID 20025615
- ↑ Cecchini G (2003.). "Function and structure of complex II of the respiratory chain". Annu Rev Biochem 72: 77–109. doi:10.1146/annurev.biochem.72.121801.161700. PMID 14527321
- ↑ Yankovskaya V., Horsefield R., Tornroth S., Luna-Chavez C., Miyoshi H., Leger C., Byrne B., Cecchini G. et al. (2003.). "Architecture of succinate dehydrogenase and reactive oxygen species generation". Science 299 (5607): 700.–704.. doi:10.1126/science.1079605. PMID 12560550
- ↑ Horsefield R, Iwata S, Byrne B (2004). "Complex II from a structural perspective". Curr. Protein Pept. Sci. 5 (2): 107–18. doi:10.2174/1389203043486847. PMID 15078221
- ↑ Ramsay RR, Steenkamp DJ, Husain M (1987). "Reactions of electron-transfer flavoprotein and electron-transfer flavoprotein: ubiquinone oxidoreductase". Biochem. J. 241 (3): 883–92. PMC 1147643. PMID 3593226. //www.ncbi.nlm.nih.gov/pmc/articles/PMC1147643/
- ↑ Zhang J, Frerman FE, Kim JJ (2006.). "Structure of electron transfer flavoprotein-ubiquinone oxidoreductase and electron transfer to the mitochondrial ubiquinone pool". Proc. Natl. Acad. Sci. U.S.A. 103 (44): 16212–7. doi:10.1073/pnas.0604567103. PMC 1637562. PMID 17050691. //www.ncbi.nlm.nih.gov/pmc/articles/PMC1637562/
- ↑ Ikeda Y, Dabrowski C, Tanaka K (25. siječnja 1983.). "Separation and properties of five distinct acyl-CoA dehydrogenases from rat liver mitochondria. Identification of a new 2-methyl branched chain acyl-CoA dehydrogenase". J. Biol. Chem. 258 (2): 1066–76. PMID 6401712. http://www.jbc.org/cgi/reprint/258/2/1066
- ↑ Ruzicka FJ, Beinert H (1977.). "A new iron-sulfur flavoprotein of the respiratory chain. A component of the fatty acid beta oxidation pathway" (PDF). J. Biol. Chem. 252 (23): 8440–5. PMID 925004. http://www.jbc.org/cgi/reprint/252/23/8440.pdf
- ↑ Ishizaki K, Larson TR, Schauer N, Fernie AR, Graham IA, Leaver CJ (2005.). "The critical role of Arabidopsis electron-transfer flavoprotein:ubiquinone oxidoreductase during dark-induced starvation". Plant Cell 17 (9): 2587–600. doi:10.1105/tpc.105.035162. PMC 1197437. PMID 16055629. //www.ncbi.nlm.nih.gov/pmc/articles/PMC1197437/
- ↑ Berry E, Guergova-Kuras M, Huang L, Crofts A (2000.). "Structure and function of cytochrome bc complexes". Annu Rev Biochem 69: 1005.–75.. doi:10.1146/annurev.biochem.69.1.1005. PMID 10966481
- ↑ Crofts AR (2004.). "The cytochrome bc1 complex: function in the context of structure". Annu. Rev. Physiol. 66: 689.–733.. doi:10.1146/annurev.physiol.66.032102.150251. PMID 14977419
- ↑ Iwata S, Lee JW, Okada K, et al. (1998.). "Complete structure of the 11-subunit bovine mitochondrial cytochrome bc1 complex". Science 281 (5373): 64.–71.. doi:10.1126/science.281.5373.64. PMID 9651245
- ↑ Trumpower BL (1990). "The protonmotive Q cycle. Energy transduction by coupling of proton translocation to electron transfer by the cytochrome bc1 complex" (PDF). J. Biol. Chem. 265 (20): 11409–12. PMID 2164001. http://www.jbc.org/cgi/reprint/265/20/11409.pdf
- ↑ Hunte C, Palsdottir H, Trumpower BL (2003). "Protonmotive pathways and mechanisms in the cytochrome bc1 complex". FEBS Lett. 545 (1): 39–46. doi:10.1016/S0014-5793(03)00391-0. PMID 12788490
- ↑ Calhoun M, Thomas J, Gennis R (1994). "The cytochrome oxidase superfamily of redox-driven proton pumps". Trends Biochem Sci 19 (8): 325–30. doi:10.1016/0968-0004(94)90071-X. PMID 7940677
- ↑ Tsukihara T, Aoyama H, Yamashita E, Tomizaki T, Yamaguchi H, Shinzawa-Itoh K, Nakashima R, Yaono R, Yoshikawa S. (1996). "The whole structure of the 13-subunit oxidized cytochrome c oxidase at 2.8 A". Science 272 (5265): 1136–44. Bibcode 1996Sci...272.1136T. doi:10.1126/science.272.5265.1136. PMID 8638158
- ↑ Yoshikawa S, Muramoto K, Shinzawa-Itoh K, et al. (2006). "Proton pumping mechanism of bovine heart cytochrome c oxidase". Biochim. Biophys. Acta 1757 (9–10): 1110–6. doi:10.1016/j.bbabio.2006.06.004. PMID 16904626
- ↑ Rasmusson AG, Soole KL, Elthon TE (2004). "Alternative NAD(P)H dehydrogenases of plant mitochondria". Annual review of plant biology 55: 23–39. doi:10.1146/annurev.arplant.55.031903.141720. PMID 15725055
- ↑ Menz RI, Day DA (1996). "Purification and characterization of a 43-kDa rotenone-insensitive NADH dehydrogenase from plant mitochondria". J. Biol. Chem. 271 (38): 23117–20. doi:10.1074/jbc.271.38.23117. PMID 8798503. http://www.jbc.org/cgi/content/full/271/38/23117
- ↑ McDonald A, Vanlerberghe G (2004). "Branched mitochondrial electron transport in the Animalia: presence of alternative oxidase in several animal phyla". IUBMB Life 56 (6): 333–41. doi:10.1080/1521-6540400000876. PMID 15370881
- ↑ Sluse FE, Jarmuszkiewicz W (1998). "Alternative oxidase in the branched mitochondrial respiratory network: an overview on structure, function, regulation, and role". Braz. J. Med. Biol. Res. 31 (6): 733–47. doi:10.1590/S0100-879X1998000600003. PMID 9698817
- ↑ Moore AL, Siedow JN (1991). "The regulation and nature of the cyanide-resistant alternative oxidase of plant mitochondria". Biochim. Biophys. Acta 1059 (2): 121–40. doi:10.1016/S0005-2728(05)80197-5. PMID 1883834
- ↑ Vanlerberghe GC, McIntosh L (1997). "Alternative oxidase: From Gene to Function". Annual Review of Plant Physiology and Plant Molecular Biology 48: 703–34. doi:10.1146/annurev.arplant.48.1.703. PMID 15012279
- ↑ Ito Y, Saisho D, Nakazono M, Tsutsumi N, Hirai A (1997). "Transcript levels of tandem-arranged alternative oxidase genes in rice are increased by low temperature". Gene 203 (2): 121–9. doi:10.1016/S0378-1119(97)00502-7. PMID 9426242
- ↑ Maxwell DP, Wang Y, McIntosh L (1999). "The alternative oxidase lowers mitochondrial reactive oxygen production in plant cells". Proc. Natl. Acad. Sci. U.S.A. 96 (14): 8271–6. doi:10.1073/pnas.96.14.8271. PMC 22224. PMID 10393984. http://www.pnas.org/cgi/content/full/96/14/8271
- ↑ Lenaz G (2001). "A critical appraisal of the mitochondrial coenzyme Q pool". FEBS Lett. 509 (2): 151–5. doi:10.1016/S0014-5793(01)03172-6. PMID 11741580
- ↑ Heinemeyer J, Braun HP, Boekema EJ, Kouril R (2007). "A structural model of the cytochrome C reductase/oxidase supercomplex from yeast mitochondria". J. Biol. Chem. 282 (16): 12240–8. doi:10.1074/jbc.M610545200. PMID 17322303
- ↑ Schägger H, Pfeiffer K (2000). "Supercomplexes in the respiratory chains of yeast and mammalian mitochondria". EMBO J. 19 (8): 1777–83. doi:10.1093/emboj/19.8.1777. PMC 302020. PMID 10775262. //www.ncbi.nlm.nih.gov/pmc/articles/PMC302020/
- ↑ Schägger H (2002). "Respiratory chain supercomplexes of mitochondria and bacteria". Biochim. Biophys. Acta 1555 (1–3): 154–9. doi:10.1016/S0005-2728(02)00271-2. PMID 12206908
- ↑ Schägger H, Pfeiffer K (2001). "The ratio of oxidative phosphorylation complexes I-V in bovine heart mitochondria and the composition of respiratory chain supercomplexes". J. Biol. Chem. 276 (41): 37861–7. doi:10.1074/jbc.M106474200. PMID 11483615. http://www.jbc.org/cgi/content/full/276/41/37861
- ↑ Gupte S, Wu ES, Hoechli L, et al. (1984). "Relationship between lateral diffusion, collision frequency, and electron transfer of mitochondrial inner membrane oxidation-reduction components". Proc. Natl. Acad. Sci. U.S.A. 81 (9): 2606–10. doi:10.1073/pnas.81.9.2606. PMC 345118. PMID 6326133. //www.ncbi.nlm.nih.gov/pmc/articles/PMC345118/
- ↑ Nealson KH (1999). "Post-Viking microbiology: new approaches, new data, new insights". Origins of life and evolution of the biosphere: the journal of the International Society for the Study of the Origin of Life 29 (1): 73–93. doi:10.1023/A:1006515817767. PMID 11536899
- ↑ Schäfer G, Engelhard M, Müller V (1999). "Bioenergetics of the Archaea". Microbiol. Mol. Biol. Rev. 63 (3): 570–620. PMC 103747. PMID 10477309. //www.ncbi.nlm.nih.gov/pmc/articles/PMC103747/
- ↑ Ingledew WJ, Poole RK (1984). "The respiratory chains of Escherichia coli". Microbiol. Rev. 48 (3): 222–71. PMC 373010. PMID 6387427. //www.ncbi.nlm.nih.gov/pmc/articles/PMC373010/
- ↑ Unden G, Bongaerts J (1997). "Alternative respiratory pathways of Escherichia coli: energetics and transcriptional regulation in response to electron acceptors". Biochim. Biophys. Acta 1320 (3): 217–34. doi:10.1016/S0005-2728(97)00034-0. PMID 9230919
- ↑ Iuchi S, Lin EC (1993.). "Adaptation of Escherichia coli to redox environments by gene expression". Mol. Microbiol. 9 (1): 9–15. doi:10.1111/j.1365-2958.1993.tb01664.x. PMID 8412675
- ↑ Calhoun MW, Oden KL, Gennis RB, de Mattos MJ, Neijssel OM (1993). "Energetic efficiency of Escherichia coli: effects of mutations in components of the aerobic respiratory chain" (PDF). J. Bacteriol. 175 (10): 3020–5. PMC 204621. PMID 8491720. http://jb.asm.org/cgi/reprint/175/10/3020.pdf
- ↑ 57,0 57,1 Boyer PD (1997). "The ATP synthase—a splendid molecular machine". Annu. Rev. Biochem. 66: 717–49. doi:10.1146/annurev.biochem.66.1.717. PMID 9242922
- ↑ Van Walraven HS, Strotmann H, Schwarz O, Rumberg B (1996). "The H+/ATP coupling ratio of the ATP synthase from thiol-modulated chloroplasts and two cyanobacterial strains is four". FEBS Lett. 379 (3): 309–13. doi:10.1016/0014-5793(95)01536-1. PMID 8603713
- ↑ Yoshida M, Muneyuki E, Hisabori T (2001). "ATP synthase—a marvellous rotary engine of the cell". Nat. Rev. Mol. Cell Biol. 2 (9): 669–77. doi:10.1038/35089509. PMID 11533724
- ↑ Schemidt RA, Qu J, Williams JR, Brusilow WS (1998). "Effects of carbon source on expression of Fo genes and on the stoichiometry of the c subunit in the F1Fo ATPase of Escherichia coli". J. Bacteriol. 180 (12): 3205–8. PMC 107823. PMID 9620972. https://www.ncbi.nlm.nih.gov/pmc/articles/PMC107823/
- ↑ Nelson N, Perzov N, Cohen A, Hagai K, Padler V, Nelson H (1 siječanj 2000). "The cellular biology of proton-motive force generation by V-ATPases". J. Exp. Biol. 203 (Pt 1): 89–95. PMID 10600677. http://jeb.biologists.org/cgi/reprint/203/1/89
- ↑ Rubinstein JL, Walker JE, Henderson R (2003). "Structure of the mitochondrial ATP synthase by electron cryomicroscopy". EMBO J. 22 (23): 6182–92. doi:10.1093/emboj/cdg608. PMC 291849. PMID 14633978. //www.ncbi.nlm.nih.gov/pmc/articles/PMC291849/
- ↑ Leslie AG, Walker JE (2000). "Structural model of F1-ATPase and the implications for rotary catalysis". Philos. Trans. R. Soc. Lond., B, Biol. Sci. 355 (1396): 465–71. doi:10.1098/rstb.2000.0588. PMC 1692760. PMID 10836500. //www.ncbi.nlm.nih.gov/pmc/articles/PMC1692760/
- ↑ Noji H, Yoshida M (2001). "The rotary machine in the cell, ATP synthase". J. Biol. Chem. 276 (3): 1665–8. doi:10.1074/jbc.R000021200. PMID 11080505. http://www.jbc.org/cgi/content/full/276/3/1665
- ↑ Capaldi R, Aggeler R (2002). "Mechanism of the F(1)F(0)-type ATP synthase, a biological rotary motor". Trends Biochem Sci 27 (3): 154–60. doi:10.1016/S0968-0004(01)02051-5. PMID 11893513
- ↑ Dimroth P, von Ballmoos C, Meier T (2006). "Catalytic and mechanical cycles in F-ATP synthases. Fourth in the Cycles Review Series". EMBO Rep 7 (3): 276–82. doi:10.1038/sj.embor.7400646. PMC 1456893. PMID 16607397. //www.ncbi.nlm.nih.gov/pmc/articles/PMC1456893/
- ↑ 67,0 67,1 Gresser MJ, Myers JA, Boyer PD (25 rujan 1982). "Catalytic site cooperativity of beef heart mitochondrial F1 adenosine triphosphatase. Correlations of initial velocity, bound intermediate, and oxygen exchange measurements with an alternating three-site model". J. Biol. Chem. 257 (20): 12030–8. PMID 6214554. http://www.jbc.org/cgi/reprint/257/20/12030
- ↑ Dimroth P (1994). "Bacterial sodium ion-coupled energetics". Antonie Van Leeuwenhoek 65 (4): 381–95. doi:10.1007/BF00872221. PMID 7832594
- ↑ Becher B, Müller V (1994). "Delta mu Na+ drives the synthesis of ATP via a delta mu Na(+)-translocating F1F0-ATP synthase in membrane vesicles of the archaeon Methanosarcina mazei Gö1". J. Bacteriol. 176 (9): 2543–50. PMC 205391. PMID 8169202. https://www.ncbi.nlm.nih.gov/pubmed/8169202
- ↑ Müller V (2004.). "An exceptional variability in the motor of archaeal A1AO ATPases: from multimeric to monomeric rotors comprising 6–13 ion binding sites". J. Bioenerg. Biomembr. 36 (1): 115.–25.. doi:10.1023/B:JOBB.0000019603.68282.04. PMID 15168615. https://link.springer.com/article/10.1023/B:JOBB.0000019603.68282.04
- ↑ 71,0 71,1 Davies K (1995.). "Oxidative stress: the paradox of aerobic life". Biochem Soc Symp 61: 1–31. PMID 8660387
- ↑ Rattan SI (2006.). "Theories of biological aging: genes, proteins, and free radicals". Free Radic. Res. 40 (12): 1230–8. doi:10.1080/10715760600911303. PMID 17090411
- ↑ Valko M, Leibfritz D, Moncol J, Cronin MT, Mazur M, Telser J (2007.). "Free radicals and antioxidants in normal physiological functions and human disease". Int. J. Biochem. Cell Biol. 39 (1): 44–84. doi:10.1016/j.biocel.2006.07.001. PMID 16978905
- ↑ Raha S, Robinson B (2000.). "Mitochondria, oxygen free radicals, disease and ageing". Trends Biochem Sci 25 (10): 502.–8.. doi:10.1016/S0968-0004(00)01674-1. PMID 11050436
- ↑ Finkel T, Holbrook NJ (2000). "Oxidants, oxidative stress and the biology of ageing". Nature 408 (6809): 239–47. doi:10.1038/35041687. PMID 11089981
- ↑ Kadenbach B, Ramzan R, Wen L, Vogt S (svibanj 2009). "New extension of the Mitchell Theory for oxidative phosphorylation in mitochondria of living organisms". Biochim. Biophys. Acta 1800 (3): 205–212. doi:10.1016/j.bbagen.2009.04.019. PMID 19409964
- ↑ Echtay KS, Roussel D, St-Pierre J, et al. (siječanj 2002). "Superoxide activates mitochondrial uncoupling proteins". Nature 415 (6867): 96–9. doi:10.1038/415096a. PMID 11780125
- ↑ Tsubaki M; Yoshikawa, Shinya (1993.). "Fourier-transform infrared study of cyanide binding to the Fea3-CuB binuclear site of bovine heart cytochrome c oxidase: implication of the redox-linked conformational change at the binuclear site". Biochemistry 32 (1): 164.–73.. doi:10.1021/bi00052a022. PMID 8380331
- ↑ Joshi S, Huang YG (1991.). "ATP synthase complex from bovine heart mitochondria: the oligomycin sensitivity conferring protein is essential for dicyclohexyl carbodiimide-sensitive ATPase". Biochim. Biophys. Acta 1067 (2): 255.–8.. doi:10.1016/0005-2736(91)90051-9. PMID 1831660
- ↑ Heytler PG (1979.). "Uncouplers of oxidative phosphorylation". Meth. Enzymol. 55: 462.–42.. doi:10.1016/0076-6879(79)55060-5. PMID 156853
- ↑ Lambert AJ, Brand MD (2004.). "Inhibitors of the quinone-binding site allow rapid superoxide production from mitochondrial NADH: ubiquinone oxidoreductase (complex I)". J. Biol. Chem. 279 (38): 39414.–20.. doi:10.1074/jbc.M406576200. PMID 15262965. http://www.jbc.org/cgi/content/full/279/38/39414
- ↑ Dervartanian DV, Veeger C. (studeni 1964). "Studies on succinate dehydrogenase. I. Spectral properties of the purified enzyme and formation of enzyme-competitive inhibitor complexes". Biochim. Biophys. Acta 92: 233–47. PMID 14249115
- ↑ Ricquier D, Bouillaud F (2000). "The uncoupling protein homologues: UCP1, UCP2, UCP3, StUCP and AtUCP". Biochem. J. 345 Pt 2: 161–79. doi:10.1042/0264-6021:3450161. PMC 1220743. PMID 10620491. //www.ncbi.nlm.nih.gov/pmc/articles/PMC1220743/
- ↑ Harden A, Young WJ. (1906). "The alcoholic ferment of yeast-juice". Proc. R. Soc. (Lond.) B (77): 405–20. doi:10.1098/rspb.1906.0029
- ↑ Kalckar HM (1974). "Origins of the concept oxidative phosphorylation". Mol. Cell. Biochem. 5 (1–2): 55–63. doi:10.1007/BF01874172. PMID 4279328
- ↑ Friedkin M, Lehninger AL. (1. travanj 1949.). "Esterification of inorganic phosphate coupled to electron transport between dihydrodiphosphopyridine nucleotide and oxygen". J. Biol. Chem. 178 (2): 611–23. PMID 18116985. http://www.jbc.org/cgi/reprint/178/2/611
- ↑ Slater EC. (1953). "Mechanism of Phosphorylation in the Respiratory Chain". Nature 172 (4387): 975–8. doi:10.1038/172975a0. PMID 13111237
- ↑ Mitchell P. (1961). "Coupling of Phosphorylation to Electron and Hydrogen Transfer by a Chemi-Osmotic type of Mechanism". Nature 191 (4784): 144–8. Bibcode 1961Natur.191..144M. doi:10.1038/191144a0. PMID 13771349
- ↑ Milton H. Saier Jr. "Peter Mitchell and the Vital Force". http://www-biology.ucsd.edu/~msaier/transport/petermitchell/MitchellFrame-1.html Pristupljeno 23. kolovoz 2007.
- ↑ Mitchell, Peter (1978). "David Keilin's Respiratory Chain Concept and Its Chemiosmotic Consequences" (Pdf). Nobel lecture. Nobel Foundation. http://nobelprize.org/nobel_prizes/chemistry/laureates/1978/mitchell-lecture.pdf Pristupljeno 21. srpanj 2007.
- ↑ Boyer PD, Cross RL, Momsen W (1973). "A new concept for energy coupling in oxidative phosphorylation based on a molecular explanation of the oxygen exchange reactions". Proc. Natl. Acad. Sci. U.S.A. 70 (10): 2837–9. doi:10.1073/pnas.70.10.2837. PMC 427120. PMID 4517936. //www.ncbi.nlm.nih.gov/pmc/articles/PMC427120/
- ↑ "The Nobel Prize in Chemistry 1997". Nobel Foundation. http://nobelprize.org/nobel_prizes/chemistry/laureates/1997/ Pristupljeno 21. srpanj 2007.
Vanjske poveznice
- (engl.)Animated diagrams illustrating oxidative phosphorylation Wiley and Co Concepts in Biochemistry
- (engl.)On-line biophysics lectures Antony Crofts, University of Illinois at Urbana-Champaign
- (engl.)ATP synthase
- (engl.)Cytochrome c
- (engl.)Cytochrome c oxidase
- (engl.)NADH dehydrogenase
- (engl.)succinate dehydrogenase
- (engl.)Coenzyme Q - cytochrome c reductase
- (engl.)cytochrome c oxidase
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||







