Sarbecovirus
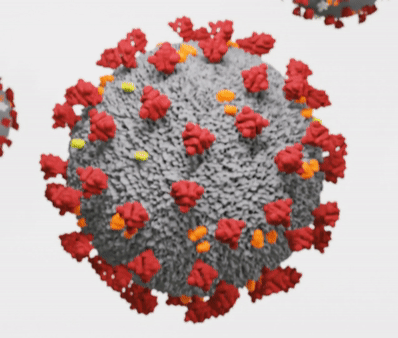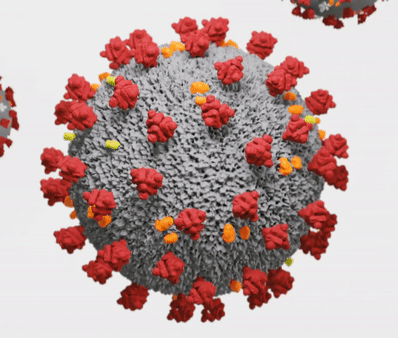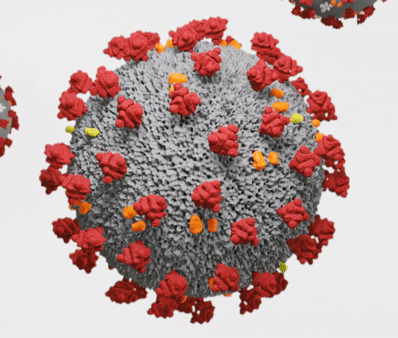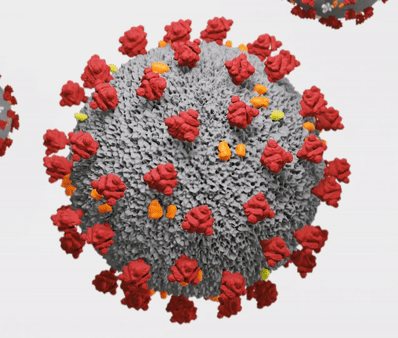
Teški akutni respiratorni sindrom koronavirus (SARSr-CoV) ili Sarbecovirus je vrsta koronavirusa sa spobnosću zaraze ljudi, šišmiša i određene vrste sisara. To je omotani jednolančani RNK virus pozitivnog osjećaja koji ulazi u stanicu domaćina vezanjem na receptor enzima 2 (ACE2) za konverziju angiotenzina. Član je roda Betacoronavirus i subgena Sarbecoronavirus.[1][2]
SARS coronavirus bio je jedan od nekoliko virusa koje je Svjetska zdravstvena organizacija (WHO) 2016. identificirala kao vjerojatni uzrok buduće epidemije u novom planu razvijenom nakon epidemije ebole za hitna istraživanja i razvoj prije i tijekom epidemije prema dijagnostici testovi, cjepiva i lijekovi. Predviđanje se obistinilo pandemijom koronavirusa 2019.-20.
Porijeklo
Šišmiši služe kao glavni rezervoar domaćina za koronavirus povezan sa SARS-om. Virus se koevoluirao u rezervoaru domaćina šišmiša tijekom dugog vremenskog razdoblja. Tek nedavno su evoluirali sojevi koronavirusa povezanih sa SARS-om i omogućili da vrsta vrsta preskoči sa slijepih miševa na ljude, kao u slučaju sojeva SARS-CoV i SARS-CoV-2. Oba ova soja potječu od jednog jedinog pretka, ali su križane vrste odvojeno skočile u ljude. SARS-CoV-2 nije izravni potomak SARS-CoV.[3][4]
Morfologija
Morfologija koronavirusa povezanih sa SARS karakteristična je za obitelj koronavirusa u cjelini. Virusi su velike pleomorfne sferne čestice s bulbous površinskim izbočenjima koje tvore koronu oko čestica u elektronskim mikrografima. Veličina virusnih čestica je u rasponu od 80–90 nm. Ovojnica virusa u elektronskim mikrografima pojavljuje se kao poseban par elektronski gustih školjki.[5][6][7]
Unutar ovojnice nalazi se nukleokapsid, koji je formiran od višestrukih kopija nukleokapsidnog (N) proteina, koji su vezani za jednolančani (približno 30 kb) genom RNK u neprekidnom RNK genomu u kontinuiranom zrncu-na- struna tipa strune. Ovojnica lipidnog dvosloja, membranski proteini i nukleokapsid štite virus kada je izvan domaćina.[8]
Vidi još
Izvori
- ↑ Virus Taxonomy: 2018 Release. International Committee on Taxonomy of Viruses (ICTV) 0. October 2018 0. Pristupljeno 13 January 2019.
- ↑ • Nepoznat parametar:
vauthors
• Nepoznat parametar:issue
• Nepoznat parametar:pmc
• Parametarpmidnije dopušten u klasijournal
• Parametartypenije dopušten u klasijournal
• Parametardatenije dopušten u klasijournal - ↑ • Nepoznat parametar:
vauthors
• Nepoznat parametar:issue
• Nepoznat parametar:pmc
• Parametarpmidnije dopušten u klasijournal
• Parametartypenije dopušten u klasijournal
• Parametardatenije dopušten u klasijournal - ↑ Coronaviridae - Figures - Positive Sense RNA Viruses - Positive Sense RNA Viruses (2011). International Committee on Taxonomy of Viruses (ICTV) 0. Pristupljeno 2020-03-06.
- ↑
• Nepoznat parametar:
vauthors - ↑ • Nepoznat parametar:
vauthors
• Nepoznat parametar:issue
• Nepoznat parametar:pmc
• Parametarpmidnije dopušten u klasijournal
• Parametartypenije dopušten u klasijournal
• Parametardatenije dopušten u klasijournal - ↑ • Nepoznat parametar:
display-authors
• Nepoznat parametar:vauthors
• Nepoznat parametar:issue
• Nepoznat parametar:pmc
• Parametartypenije dopušten u klasijournal
• Parametardatenije dopušten u klasijournal
• Parametarpmidnije dopušten u klasijournal - ↑ • Nepoznat parametar:
display-authors
• Nepoznat parametar:vauthors
• Nepoznat parametar:issue
• Nepoznat parametar:pmc
• Parametartypenije dopušten u klasijournal
• Parametardatenije dopušten u klasijournal
• Parametarpmidnije dopušten u klasijournal